Contenido principal
Alfa 46
El gigantesco páramo blanco de la Antártida tiene como habitante a uno de los mayores telescopios del mundo: el Ice- Cube. Este hueco de un kilómetro cúbico excavado a 2.500 metros bajo el hielo consta de unos 5.000 sensores y cumple diez años ahora.
Tras la secuenciación del genoma humano y del proteoma, el interactoma abre hoy las puertas al estudio del comportamiento celular desde otra perspectiva, la cual permitirá comprender enfermedades y hallar nuevos tratamientos mediante la aplicación de nuevas técnicas de nanotecnología y biomedicina.
Además incluimos un contenido dedicado a la luz ultravioleta, invisible a nuestros ojos, pero peligrosa para nuestra piel. La comunicación con la ciudadanía, la transparencia y la necesaria independencia del CSN son algunos de los asuntos que aborda en sus respuestas la consejera Pilar Lucio, protagonista de la entrevista de este trimestre.
Interactoma, la red social de las proteínas

Si hace 20 años, el Proyecto Genoma Humano anunció la secuencia completa de nuestro ADN, hoy la ciencia ha ido más allá. De él se ha obtenido un mapa aún más complejo, el atlas que desvela cómo todas nuestras proteínas se relacionan dentro de la célula. Es lo que se conoce como interactoma, y abre las puertas al estudio de enfermedades y terapias de una forma distinta. Acelerará sus avances, los de la bio y la nanotecnología, los de la biología del desarrollo y los estudios sobre la evolución. Sus resultados se comparten en una inmensa base de datos que condensa el saber de décadas de investigación.
Texto: Mar de Miguel | Periodista científica
En septiembre de 2011, la Universidad de la Ciudad de Ho Chi Minh (antigua Saigón, en Vietnam) presentó un puzle de 551.232 piezas que formaban una enorme flor de loto. Para completarlo, colaboraron 1.600 estudiantes que emplearon 17 horas. Hay que reconocer que reordenar cada trocito de una imagen fragmentada tantas veces resulta laborioso. Sin embargo, partieron de un molde conocido, reconstruido con piezas únicas que pudieron ver, tocar y encajar en una posición concreta. Cuando los científicos hacen esto mismo, pero descienden a nivel molecular, la cosa se complica.
El 15 de febrero de 2001, hace 20 años, las revistas Nature y Science publicaron el primer borrador de nuestro genoma. El ensamblaje completo de las piezas de nuestro ADN llegó el 24 de abril de 2003, justo a tiempo para celebrar el 50 aniversario de la publicación de su estructura, descubierta por James Watson y Francis Crick.
Los investigadores del Proyecto Genoma Humano, liderado por los Institutos Nacionales de Salud de EEUU (NIH, por sus siglas en inglés) junto a 20 instituciones de siete países distintos, tardaron 13 años en completar este rompecabezas genético. A ciegas y sin un modelo previo en el que fijarse, anotaron y acoplaron ordenadamente los 3.200 millones de pares de bases de nuestros 23 pares de cromosomas.
La historia se repite ahora, de la mano del Instituto del Cáncer DanaFarber de Boston y la Universidad de Harvard (EE. UU.) con el aún más complejo proyecto del Interactoma Humano. Ya no se trata sólo de saber quién es quién o dónde está, sino de entender sus relaciones. Quién habla, quién escucha, a quién se dice qué, quién impide la comunicación de un grupo o quién la favorece e invita a otros a la reunión. Los protagonistas son las proteínas. Las salas para sus contactos son las células y sus compartimentos. El gran congreso proteico tiene lugar en el cuerpo humano.
“Hemos mapeado el interactoma humano de interacciones moleculares entre proteínas. También existen mapas de asociación genética, que recogen las interacciones entre los genes”, señala Javier De Las Rivas, coautor del trabajo y director del Grupo de Investigación en Bioinformática y Genómica Funcional del Centro de Investigación del Cáncer (CSIC y Universidad de Salamanca). El estudio, publicado en la revista Nature, está dirigido por Marc Vidal, director del Instituto del Cáncer Dana-Farber de Boston y catedrático de Genética de la Escuela de Medicina Harvard, en colaboración con otros 30 grupos de investigación de nueve países.
Un proyecto colosal
Para obtener el interactoma humano, primero clonaron los 20.000 genes que generan todas las proteínas de nuestra especie. De ellos, obtuvieron con éxito 17.500 proteínas. Este trabajo formó parte de un proyecto anterior llamado ORFeoma. Aunque existen ORFeomas de bacterias, el laboratorio de Vidal en Boston es el único en el mundo que lo desarrolló en humanos.
Una vez conseguidas las 17.500 proteínas, para saber cuáles de ellas interaccionan, estudiaron el modo en que se acoplan. Las proteínas están formadas por cadenas de aminoácidos plegadas que se unen entre sí y se repliegan aún más. El resultado es una estructura tridimensional que deja un espacio de unión a otras proteínas. Dos proteínas distintas que interaccionan se unen por estos puntos, como se observa mediante microscopía electrónica de muy alta resolución y modelos 3D.
Para lograr el interactoma de las 17.500 proteínas, aplicaron la tecnología de interacción molecular de doble híbrido. La técnica consiste en medir las interacciones entre pares de proteínas. Para ello, realizaron millones de experimentos binarios. Con ellos probaron si una proteína conectaba con otra, después con una tercera y así hasta 17.500 veces.
Pero si obtener proteínas es complejo, asegurar que las interacciones positivas realmente lo son dispara el número de experimentos. “Las interacciones binarias de dos proteínas (una proteína A contra una proteína B y la B contra la A) se testan tres veces”, explica De Las Rivas. Esto supone seis experimentos por interacción para 17.500 proteínas, es decir, un número colosal de pruebas, que además aplicaron con tres técnicas distintas. “Hemos testado 153 millones de interacciones”, añade. De todas ellas, sólo 53.000 fueron positivas.
Cabe preguntarse a cuánto asciende la factura de un trabajo de esta envergadura. Han sido varios millones de dólares, financiados por el NIH estadounidense y por las entidades científicas que han colaborado, añadiendo los fondos de 15 proyectos y becas.
Interactomas contra la covid-19
Las interacciones de proteínas humanas no son las únicas descritas. También existen interactomas de bacterias, virus y de varios modelos animales. Las bases de datos de diferentes especies se consultan cuando no se conoce la función de una proteína o su interacción con otras moléculas, lo que abre el abanico a estudios evolutivos de carácter molecular. Además, el universo de las interacciones de moléculas puede aumentar su rango. Si no solo contemplamos relaciones binarias sino que vemos cómo se comporta un grupo de proteínas, construiríamos una red multidimensional compleja. Ésta no se queda en lo que pasa en el interior de una célula, sino que desengrana la maquinaria de todo un organismo.
Los interactomas no han hecho más que empezar y aumentarán en un futuro porque nos ayudarán a combatir distintas enfermedades. Un buen ejemplo de ello es el interactoma del SARS-CoV-2, el virus causante de la pandemia que asola el mundo actualmente. Apenas un año después de su aparición, se ha propagado por 192 países y se ha cobrado la vida de dos millones y medio de personas. Laboratorios y farmacéuticas, como Pfizer, Moderna o AstraZeneca, disponen de vacunas prometedoras que ya se aplican en varias regiones del mundo, al mismo tiempo que el virus desarrolla mutaciones capaces de escapar de ellas.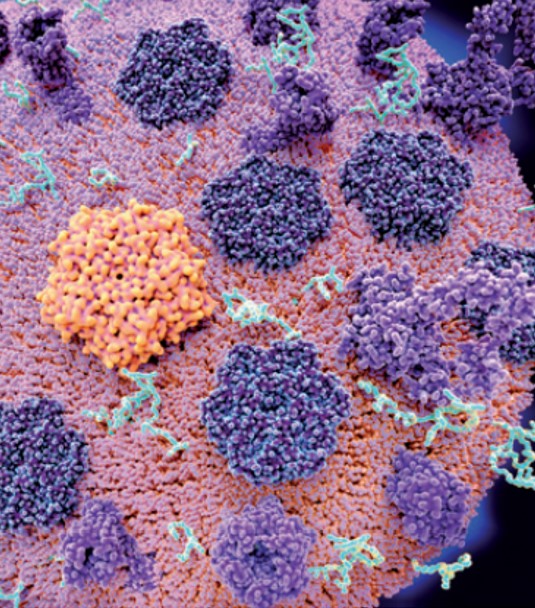
El reto está en alcanzar a tiempo la inmunización de un 70 % de la población mundial, como indica la Organización Mundial de la Salud (OMS), para detener contagios y muertes. Por otro lado, se avanza en el tratamiento de la sintomatología, pero aún no disponemos de un fármaco que elimine las partículas víricas de nuestro organismo, tal como un antibiótico lo haría ante una infección bacteriana. La clave del éxito está en conocer el virus a fondo, en identificar las piezas de su rompecabezas genético y proteico, en entender su funcionamiento para desarmarlo.
En eso trabaja el virólogo español Adolfo García-Sastre, catedrático de Microbiología y director del Instituto de Salud Global y Patógenos Emergentes de la Escuela de Medicina Icahn en Mount Sinai, Nueva York (EEUU). Él y sus colaboradores de la Universidad de California en San Francisco (EEUU), del Instituto Pasteur (Francia) y otros 30 laboratorios estadounidenses y de Reino Unido, han publicado en la revista Nature el mapa de interacción de las proteínas del virus SARS-CoV-2 con las proteínas humanas. “Espero que este mapa sirva para acelerar los estudios sobre la biología del SARS-CoV-2”, dice García-Sastre.
Su objetivo es encontrar candidatos a fármacos, moléculas que nos ayuden a erradicar el virus, su tratamiento o el control de la pandemia. En el interactoma recién descrito ya destaca uno, la plitidepsina, un potente antiviral que bloquea la proteína eEF1A del huésped, lo que impide la replicación del SARS-CoV-2 en el organismo. Lo publica el científico burgalés en otro artículo de Science tras un ensayo preclínico en ratones.
“Es un inhibidor muy potente. Pero se necesita administrar de modo intravenoso, lo cual limita su uso si se demuestra que funciona en pacientes. Lo ideal sería un medicamento oral”, comenta García-Sastre. Este medicamento, desarrollado para otros usos por la farmacéutica española PharmaMar, se prueba en ensayos clínicos contra la covid-19. Se conoce como Aplidin, ya que procede de la ascidia Aplidium albicans, el animal marino que le da nombre.
Gracias al interactoma del SARS-CoV2 en células humanas, los científicos fijan la mirada en fármacos que ya existían. La ventaja es clara. De funcionar contra el coronavirus servirían de atajo, pues no parten de cero en su estudio y algunos ya están aprobados por distintas agencias de medicamentos. En esta revisión, GarcíaSastre y sus colegas agrupan los fármacos en dos tipos, los que inhiben la traducción de ARNm y los que están relacionados con los receptores sigma-1 y sigma-2 del retículo endoplasmático de las células del huésped: “Nos hemos centrado en los inhibidores de la traducción de ARNm, como la plitidepsina. Pero también estamos trabajando en paralelo con los reguladores de receptores sigma. Aún no tenemos un buen candidato clínico, pero no los hemos descartado todavía”, destaca el virólogo.
Con respecto a esto, un estudio preliminar publicado en JAMA, la revista de la Asociación Médica de Estados Unidos, hace hincapié en la propuesta de la Facultad de Medicina de la Universidad de Washington en San Luis (EE. UU.). Este equipo sugiere el uso del antidepresivo fluvoxamina para evitar las infecciones graves de covid-19 y minimizar los ingresos hospitalarios. El compuesto tiene una alta afinidad por el receptor sigma-1 y aumenta los niveles de serotonina en el cerebro, por lo que se utiliza para el tratamiento de la depresión o del trastorno obsesivo-compulsivo (TOC). En nuestro país, la fluvoxamina está aprobada por la Agencia Española del Medicamento bajo varios nombres comerciales.
Dianas terapéuticas para eliminar el virus
“Si te conoces a ti mismo, pero no al enemigo, por cada victoria que ganes también sufrirás una derrota”. La cita es de El Arte de la Guerra, del militar de la china antigua Sun Tzu. Aplicado al virus causante de la covid-19, desconocido hasta ahora, la comunidad científica se esfuerza en entender cómo infecta y evitar la enfermedad aislándolo, secuenciándolo y analizando sus proteínas e interacciones.
El SARS-CoV-2 es un patógeno esférico de entre 50 y 140 nanómetros de diámetro. Su estructura es sencilla. Consta de tres capas, una nucleocápside central con material genético y proteínas N, y una envoltura lipoproteica (con proteínas E y M) sobre la que se asienta una corona de espículas (o proteínas S). Su genoma (ARN+) tiene 29.891 nucleótidos con los que genera 9.860 aminoácidos y 27 proteínas.
Para la infección, las proteínas de la corona entran en contacto con la célula del huésped. Por eso, la interacción más estudiada es la de la proteína S viral y el receptor ACE2 de las células humanas, la llave de entrada del virus. Todas las vacunas se centran en esa proteína S, para que nuestro organismo la reconozca y generemos inmunidad.
Sin embargo, el virus muta y las vacunas pueden quedar obsoletas. De ahí la importancia de conocer el interactoma humano, el del SARS-CoV-2 o el de otros virus emparentados que den pistas sobre el funcionamiento de sus proteínas. En otro trabajo de la revista Science, García-Sastre e investigadores de 44 laboratorios de cinco países amplían el interactoma del SARSCoV-2 en células humanas y aportan los interactomas del SARSCoV-1 y del MERS-CoV en el huésped.
Este estudio destaca la interacción de la proteína del virus ORF9b con nuestra proteína Tom70, una conexión que ocurre de manera similar en SARSCoV-1 y en SARSCoV-2. El trabajo menciona este acoplamiento como posible diana terapéutica. “Tom70 es una proteína mitocondrial y la mitocondria tiene un papel fundamental en la respuesta antiviral innata. Nuestra hipótesis es que ORF9b interacciona con esta proteína mitocondrial para desarmar la defensa antiviral que nos proporciona la mitocondria”, explica García-Sastre.
“Si encontrásemos cómo inhibir esta interacción y, si nuestra hipótesis es correcta, restauraríamos la respuesta celular antiviral, lo que resultaría en la eliminación del virus en nuestro cuerpo”, afirma. El investigador, interesado en la respuesta antiviral innata, destaca otra relación interesante del interactoma, la de las proteínas Nup98 y Orf6, implicada en la forma en la que el virus previene la acción del interferón (responsable de la activación del sistema inmune), como también ha publicado en la revista PNAS.

 Descarga la revista completa
Descarga la revista completa